Wir bedanken uns sehr herzlich bei unseren Sponsoren für die Unterstützung!


EFB Newsletter November 2019
Thursday, 21 November 2019 05:01

 |
|
|
||
|
Deadline for Earlybird submissions: Friday 13th December.
|
||
|
|
|
|
|
Please see below the future events planned for EFB members:
|
 |
|
|
 |
Save the Dates! |
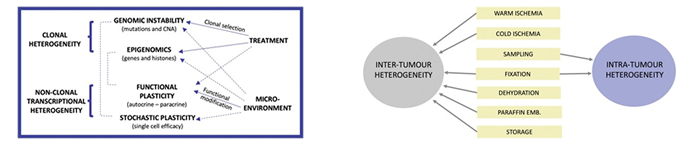 |
EFB Journal New BiotechnologyPre-analytics and tumor heterogeneity
When dealing with pre-analytics for tissues, it is often that case that tissue heterogeneity, and particularly tumor heterogeneity, is not taken into account as a preliminary condition for obtaining reproducible results in molecular analysis at the diagnostics and clinical research levels. It is well known that when sampling tumor tissues in different areas, for example the border or the central area of the tumor, different genes are expressed and, due to polyclonality in most tumors, different areas can have different DNA and epigenetic alterations. For this reason, it is extremely important to establish and standardize specific tissue sampling protocols for molecular extraction as well as in situ molecular methods. A correct approach to heterogeneity is the basis for a more reproducible and exchangeable type of molecular analysis that can provide useful information at the prognostic and predictive levels. Heterogeneity should also be taken into consideration during cancer treatment, since therapy modifies the clonal composition of tumors. Here, the different types of tumor heterogeneity and the improper pre-analytical conditions in tissue processing that can generate heterogeneous artefacts are described.
|
|
 |
SCI Event26th SCI Annual Review Meeting – Catalysis and Enabling Technologies for Synthesis
The 26th Annual Review Meeting - Catalysis and Enabling Technologies for Synthesis will address key areas of contemporary synthetic organic chemistry. An exciting line-up of leading early career academics will deliver a series of review lectures covering topics such as photoredox catalysis, biocatalysis, chemoinformatics, and automated reaction optimisation.
|
|
 |
AFOBInternational Conference on Biomaterial-Based Therapeutic Engineering and Regenerative Medicine |
|
 |
ÖGMBTMicrobe-Assisted Crop Production - Opportunities, Challenges & Needs (miCROPe 2019) |
|
EFB Member Events
organised by AFOB
13 − 14 December 2019
Gunupur − India
organised by AFOB
9 − 11 February 2020
Yogyakarta − Indonesia
organised by SCI
18 − 19 February 2020
Manchester − UK
organised by SCI
24 − 25 February 2020
Cambridge − UK
Open letter to the Austrian National Bank
Thursday, 21 November 2019 04:24The ÖGMBT together with 50 other scientific societies under the umbrella of the Federation of Austrian Scientific Societies (VWGÖ), which represents 28,599 scientists in Austria, protested against the restriction of the support of the Jubilee Fund of the Austrian National Bank. We will stay tuned and report on it.



FEBS Newsletter - November 2019
Thursday, 07 November 2019 03:52Find photos, reports and updates on a range of FEBS activities, including FEBS Council election outcomes, a round-up of the Krakow FEBS Congress, a feature on FEBS member Societies, and a first look at FEBS events lined up for 2020.
Registration and abstract submission for the 45th FEBS Congress
are now open!
Join us in the vibrant city of Ljubljana in July 2020 for inspiration, learning
and discussion across the spectrum of molecular and cellular life sciences.
Find out more on the FEBS Congress website:
Home ● Welcome ● Plenary Lectures ● Programme Overview ● Symposia ● Call for Abstracts ● Registration ● Young Scientists' Forum ● FEBS Bursaries ● Ljubljana
Key upcoming deadlines:
FEBS Young Scientists' Forum: December 15, 2019
Abstract Submission: March 5, 2020
FEBS Bursaries: March 5, 2020

Apply to the 2020 FEBS Young Scientists' Forum (YSF)
by December 15, 2019
2020 will be the 20th anniversary of this special pre-Congress meeting
for postdocs and PhD students!
Start your 2020 FEBS Congress experience with this smaller gathering in Lovran on the Croatian coast, where you can present your work, pick up career and skills tips, and get to know other early-career researchers from across the FEBS area. Those selected to take part have their participation at the YSF and the ensuing Congress supported by FEBS YSF grants. Find all the details in the YSF section of the Congress website:

The list of FEBS Advanced Courses for 2020 is now out!
FEBS-supported events within the Advanced Courses programme include lecture courses, practical courses, workshops and ‘special meetings’, and offer training,
learning and networking in focused research fields.
See the new list for 2020 on the FEBS website, together with course summaries.
FEBS Youth Travel Fund grants are available for all events to assist
participation of early-career researchers.
Images above are from the FEBS Advanced Courses 'Advanced methods in macromolecular
crystallization IX', and 'Microspectroscopy: functional imaging of biological systems'.

Have you seen the 2019 FEBS Press Special Issues?
Check out the review content from experts in...
The FEBS Journal:
ER Stress in Disease | Extracellular matrix in health and disease | Single-cell analyses
FEBS Letters:
Krakow Special Issue | The Golgi
and Molecular Oncology:
Noncoding RNAs: Dark matter of the genome and cancer | Boosting the social impact of innovative cancer research – towards a mission‐oriented approach to cancer

EFB Newsletter October 2019
Friday, 01 November 2019 01:01

 |
|
|
|
 |
Hans Clevers is group leader at the Hubrecht Institute for Developmental Biology and Stem Cell Research and at the Princess Máxima Center for Pediatric Oncology. He is also professor of Molecular Genetics at the University Medical Center Utrecht and Utrecht University and Oncode Investigator.
The Clevers group has pioneered methods to study the molecular mechanisms of tissue development and cancer of various organs. |
|
|
|
Four short talks will be included in each symposium in the scientific programme. At least half will be selected from abstracts submitted from outside Europe. To increase your chance of being selected, register now and submit your abstract immediately. |
|
|
|
|
 |
IBS2020
The International Union of Pure and Applied Chemistry (IUPAC) initiated the International Biotechnology Symposium (IBS) in Rome in 1960. Its aim was to highlight the role of chemistry in life science and biotechnology. The IBS Congress was held every 4 years, rotating between different continents. It soon became acknowledged internationally as the prime biotechnology conference. Since 2008, the IBS series has been convened biennially to address the unprecedented progress of biotechnology. The 19th IBS (IBS 2020) will be held in conjunction with the 19th European Congress on Biotechnology (ECB 2020) in Maastricht, the Netherlands from 28th June – July 1st.
|
|
 |
EppendorfStem Cell Community Day 2019
Bringing the global stem cell research community together
Following up on two successful events on recent advances in the stem cell field, Eppendorf is hosting the Stem Cell Community Day 2019 on November 21 in Lisbon, Portugal. We invite all researchers to discuss challenges, opportunities, and achievements in stem cell bioprocessing with a special focus on controlled cultivation in stirred-tank bioreactors. Check out our website as we update the program on a regular basis. |
|
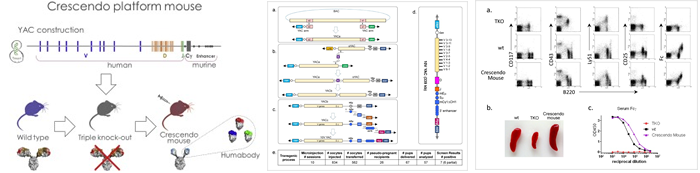 |
EFB Journal New BiotechnologyDiverse Human VH antibody fragments with bio-therapeutic properties from the Crescendo Mouse
We describe the ‘Crescendo Mouse’, a human VH transgenic platform combining an engineered heavy chain locus with diverse human heavy chain V, D and J genes, a modified mouse Cγ1 gene and complete 3’ regulatory region, in a triple knock-out (TKO) mouse background devoid of endogenous immunoglobulin expression. The addition of the engineered heavy chain locus to the TKO mouse restored B cell development, giving rise to functional B cells that responded to immunization with a diverse response that comprised entirely ‘heavy chain only’ antibodies. Heavy chain variable (VH) domain libraries were rapidly mined using phage display technology, yielding diverse high-affinity human VH that had undergone somatic hypermutation, lacked aggregation and showed enhanced expression in E. coli. The Crescendo Mouse produces human VH fragments, or Humabody® VH, with excellent bio-therapeutic potential, as exemplified here by the generation of antagonistic Humabody® VH specific for human IL17A and IL17RA.
|
|
 |
|
 |
SCI EventMIBio 2019: Stability of Biopharmaceuticals – From molecular interactions to successful products
MIBio 2019 will focus on discussing challenges linked to novel product modalities such as RNA based products or fusion proteins as well as strategies to overcome these challenges. The talks will particularly focus on protein-protein interactions, formulation, drug product development and competitive product profiles.
|
|
 |
Industry Skills Certificate
The Royal Society of Biology has developed an Industry Skills Certificate. This new initiative is designed to support the employability of scientists in industry. With extensive research and input from our Employer Advisory Group (EAG), we have created a bespoke suite of online and in-person training courses that are aimed to enhance the experience, skill set and knowledge of scientists.
|
|
|
The ÖGMBT has a very wide international network and is a member of several relevant scientific societies like EFB. ÖGMBT members are automatically members of these societies and are entitled many benefits such as 10% member discount for EFB organized congresses. Current CALLS and Deadlines are published on our webpage and are sent to our members within the ÖGMBT newsletter.
FEMS - Get Involved bulletin - October
Friday, 01 November 2019 12:14The FEMS Get involved bulletin is out! Read the October issue with microbiology research, events and calls. We want to highlight especially the open call for nominations for FEMS-Jensen Award. This Award is for young outstanding microbiology researchers and comprises a fellowhsip for at least half a year spending at an outstanding research laboratory and up to 10.000 EUR.
Content
- Join the volunteer translation team
- Building Communities - Lotte Nielsen
- Goodbye Interns
- Nominations open for FEMS-Jensen Award
- 50 Editors waive fee for grants
- Join the AcademicLabs' network
- EMA public consultation
- FEMS MEMBERS Section
The ÖGMBT has a very wide international network and is a member of several relevant scientific societies like FEMS. ÖGMBT members are automatically members of these societies and are entitled to benefits such as travel grants, short-term and long-term fellowships, Awards etc. Current CALLS and Deadlines are published on our webpage and are sent to our members within the ÖGMBT newsletter.
Wenn die Biologie rechnet
Thursday, 12 July 2018 09:36In allen Bereichen der Biowissenschaften hat man es mit stetig größer werdenden Datenmengen zu tun. Entsprechend anspruchsvoll wird die – traditionell der Informatik zukommende – Aufgabe, sie im Hinblick auf eine biologische Fragestellung zu analysieren. An dieser Schnittstelle hat sich in den vergangenen Jahrzehnten eine Disziplin etabliert, die in beiden Welten gleichermaßen zu Hause ist: die Bioinformatik. Eine ihrer ursprünglichsten Aufgaben ist dabei die Auswertung von Daten, die aus Hochdurchsatz-Sequenzierungs-Experimenten stammen. „Mit der heute am weitesten verbreiteten Illumina-Technologie erhält man Bruchstücke von 125 bis 150 Basen, aber davon 100 Millionen oder mehr“, erzählt Heinz Himmelbauer, Professor für Bioinformatik an der Universität für Bodenkultur (BOKU) Wien. Aus diesem Datenmaterial müsse das zugrunde liegende Genom erst einmal rekonstruiert werden. Die Herausforderungen bei dieser Aufgabe sind vielfältig: Daten können durch Sequenzierfehler verrauscht sein, zudem lassen sich nicht alle Bereiche des Genoms gleichermaßen einfach sequenzieren; je nach Region liegen daher unterschiedliche Mengen an Daten vor. Besondere Probleme bereiten repetitive Sequenzen, die noch dazu über weit voneinander entfernte Abschnitte des Genoms verstreut sein können.
Hat man einmal die Abfolge der Basenpaare in einem bestimmten Genom entziffert, stellt sich im nächsten Schritt die Aufgabe, zu bestimmen, wo sich darin die Gene befinden, also jene Abschnitte, die tatsächlich exprimiert werden. „Man benutzt dazu Erkennungssequenzen, die auch die Proteinmaschinerie in der Zelle dazu verwendet, an der richtigen Stelle abzulesen“, schildert Himmelbauer das Prinzip, nach dem man dabei vorgeht. Himmelbauers Gruppe hat zum Beispiel daran mitgewirkt, das Genom der Zuckerrübe zu rekonstruieren. „Von 27.000 von der Bioinformatik prognostizierten Genen konnten mehr als die Hälfte experimentell bestätigt werden“, sagt der Forscher.
Vom Genom zum Metagenom
Noch komplexer ist die Situation, wenn man nicht einen einzelnen Organismus, sondern eine ganze Gemeinschaft von Arten in ihrer natürlichen Umgebung betrachtet, etwa Mikroorganis-men, die in einer Pflanze oder in deren Wurzelbereich leben. Mit derartigen Interaktionen beschäftigt sich Günter Brader vom Kompetenzbereich „Bioresources“ des Austrian Institute of Technology (AIT): „In unserer Forschung gibt es zwei Hauptthemen: Welche Faktoren führen zur Ausbildung einer Krankheit? Und wie können Mikroorganismen der Pflanze nützen?“ Brader ist nicht selbst Bioinformatiker, sondern benutzt die Leistungen der Bioinformatik als Instrument. Gerade bei nah verwandten Stämmen ist es aber nicht einfach, in der mikrobiellen Gemeinschaft genetische Information bestimmten Organismen zuzuordnen.
Noch schwieriger ist es aber herauszufinden, was die gefundenen Gene funktionell bedeuten, also z. B. welche Metaboliten produziert werden. Zur Beantwortung solcher Fragen ist es wichtig, dass die Bioinformatik gut mit den im Labor beschäftigten Biowissenschaftlern zusammenarbeitet. „Bei Bacillus-Stämmen wissen wir durch biochemische Untersuchungen schon viel über den Metabolismus und die Proteine, die dabei eine Rolle spielen, bei anderen Organismen ist noch viel weniger Information vorhanden“, erklärt Brader.
Der Stammbaum des Lebens
Arndt von Haeseler ist Leiter des CIBIV (Center for Integrative Bioinformatics Vienna) an den Max F. Perutz Laboratories (deren wissenschaftlicher Leiter er vor kurzem wurde): „Das CIBIV wurde 2005 von Universität Wien und Meduni Wien gegründet, mit dem Ziel, die Forschungseinrichtungen am Vienna Biocenter mit Modellbildung aus der Bioinformatik anzureichern“, erzählt der Forscher. Am CIBIV verfolgt man zwei verschiedene Vor-stoßrichtungen: Zum einen werden Daten aus molekularbiologi-schen Hochdurchsatzexperimenten analysiert. „Am Campus gibt es sehr viel Grundlagenforschung, die Fragen aufwirft, die nicht Standard sind. Wir helfen mit, diese zu analysieren", erzählt von Haeseler. Dennoch will er diese Arbeit nicht als Service verstanden wissen: „Wir entwickeln neue Analysemethoden und bilden dabei eine Brücke zur ‚reinen Mathematik‘. Das hat den Charakter einer echten Forschungskooperation, die für beide Seiten zu vielen guten Publikationen führt.“
Zum anderen versucht man am CIBIV anhand eines Vergleichs von DNA-Sequenzen evolutionäre Stammbäume zu rekonstruieren. „Dabei handelt es sich um eine Optimierungsaufgabe, bei der man den am besten mit den Daten zu vereinbarenden Baum finden will“, erklärt von Haeseler. Ultimatives Ziel ist die Rekonstruktion eines riesigen Stammbaums, in den Sequenzdaten zu allen Spezies eingeflossen sind, die man kennt. Erschwert wird das durch die Möglichkeit des horizontalen Gentransfers bei ein-fachen Lebensformen: „Je weiter man im Stammbaum zurückgeht, desto mehr ähnelt dieser eher einem Netzwerk“, so von Haeseler.
Die beschriebenen Gruppen sind nur einige Beispiele von Trägern und Anwendern bioinformatischen Wissens. Wichtige Ressourcen findet man auch in der von Thomas Rattei geleiteten „Division of Computational Systems Biology” der Universität Wien. Hier interessiert man sich dafür, auf der Grundlage von bioanalytischen Daten großen Maßstabs ein Verständnis für biologische Systeme zu gewinnen – von einzelnen Arten bis zu ganzen Ökosystemen. Zusätzlich steht auch für die Bioinformatik mit dem Vienna Scientific Cluster Research Center eine universitätsübergreifende Infrastruktur auf dem Gebiet des High Performance Computing zur Verfügung.
„Broad Consent“, nicht „Blanket Consent“
Thursday, 12 July 2018 09:21Der grundsätzliche Zugang von Forschungseinrichtungen zu Beständen bezogener Daten bedeutet nicht, dass nun alles erlaubt ist.
Nach intensiven Debatten beschloss der Nationalrat am 20. April das „Datenschutzanpassungsgesetz Wissenschaft und Forschung“ zur Umsetzung der Datenschutzgrundverordnung (DSGVO) der Europäischen Union. Grob gesprochen, eröffnet das Gesetz wissenschaftlichen Einrichtungen inklusive der Forschungs- sowie Entwicklungsabteilungen von Pharmaunternehmen den rechtlich durchsetzbaren Zugriff auf bestimmte Bestände personenbezogener Daten, die aufgrund von Bundesgesetzen geführt werden. Damit wird nicht zuletzt die sogenannte „Registerforschung“ erleichtert. Überdies hat das Gesetz das Ziel, die Rechtssicherheit bei der Arbeit mit Biobanken zu erhöhen.
Ob sich diese Erwartungen erfüllen werden, ist derzeit noch schwer abzuschätzen, konstatiert Kurt Zatloukal, Mitglied der ÖGMBT, Vorstand des Diagnostik- und Forschungszentrums für Molekulare Biomedizin an der Medizinischen Universität Graz und Direktor des österreichischen Knotens der europäischen Biobankenforschungsinfrastruktur BBMRI-ERIC. Die DSGVO ermögliche den Mitgliedsstaaten, etliche der in ihr enthaltenen Bestimmungen zu spezifizieren. Und das sei differenziert zu beurteilen: „Der große Mehrwert der DSGVO war ja, ein einheitliches Rechtssystem für Europa zu schaffen und damit auch ein sicheres Umfeld zu bieten, das den Austausch von Daten erleichtert. Weichen die Bestimmungen in den Nationalstaaten zu stark voneinander ab, könnte dieser Mehrwert zumindest teilweise wieder verloren gehen.“
Inhaltlich gehen die neuen Bestimmungen laut Zatloukal in die richtige Richtung. Sehr wichtig sei etwa das Prinzip des „Broad Consent“. Im Wesentlichen besagt dieses: Ein Patient stimmt zu, dass seine (anonymisierten bzw. pseudonymisierten) Daten nicht nur für ein bestimmtes Forschungsvorhaben zur Verfügung stehen, sondern auch für sich daraus eventuell ergebende weitere Untersuchungen. Zatloukal zufolge sind weit über 90 Prozent der Patienten bereit, Proben und Daten der medizinischen Forschung zur Verfügung zu stellen, wenn sie um Zustimmung ersucht werden. Sie haben jedoch kein Interesse, diesbezüglich immer wieder kontaktiert zu werden: „Wenn jemand das dritte oder vierte Mal gefragt wird, kann es sein, dass er sagt: Vielen Dank, aber jetzt reicht es mir.“ Insofern sei der „Broad Consent“ wohl auch im Interesse der Patienten. Und Zatloukal fügt hinzu: „Broad Consent heißt nicht Blanket Consent. Man darf also nicht alles tun, ist aber zumindest in einer bestimmten Richtung frei.“ Das sei notwendig. Denn in der Grundlagenforschung könne ein Wissenschaftler am Beginn eines Vorhabens oft nicht sagen, welche Fragestellungen und weiteren Analysen sich ergeben werden.
Allerdings ist es nicht immer einfach, die Zustimmung des Patienten einzuho-len. Vom bürokratischen Aufwand abgese-hen, kann es vorkommen, dass ein Patient krankheitsbedingt nicht in der Lage ist, die Verwendung seiner Daten zu erlauben. Dies betrifft etwa den Bereich der Intensivmedizin sowie die Erforschung neurodegenerativer Erkrankungen wie Demenz. Die Nutzung derartiger Patientendaten generell zu verbieten, hieße laut Zatloukal, „bestimmte Krankheitsbilder faktisch von der Forschung abzuschneiden. Wichtig ist auch zu unterscheiden, dass die Forschung an biologischen Proben oder Daten im Labor etwas grundsätzlich anderes ist als das Testen eines neuen Medikaments am Patienten selbst“.
Dass auch Pharmaunternehmen unter bestimmten Voraussetzungen personenbezogene Daten für Forschungszwecke nutzen dürfen, ist Zatloukal zufolge unverzichtbar. Anders sei die Entwicklung neuer Diagnostika und Arzneimittel nicht möglich: „Auf europäischer Ebene differenziert man deshalb nicht zwischen akademischer und industrieller Forschung, und der Forschungsbegriff ist bewusst sehr weit gehalten“.
Nicht zu unterschätzen sind laut Zatloukal die Herausforderungen bezüglich der personalisierten Medizin. Deren Charakteristikum bestehe nun einmal darin, „Erkrankungen so detailliert zu beschreiben, dass die Merkmale einzelner Patienten im Vordergrund stehen. Die Forschung im Kontext der personalisierten Medizin kommt daher an der Notwendigkeit, mit identifizierbaren Daten zu arbeiten, nicht vorbei. Anonymisieren ist daher für Forschungen im Bereich der personalisierten Medizin oft nicht machbar“.
Schutzwürdige Daten
Eine ähnliche Position vertritt der Fachverband der Chemischen Industrie Österreichs (FCIO). Der zuständige Referent, Franz Latzko, verweist auf die Unverzichtbarkeit klinischer Studien, um „die Sicherheit und Wirksamkeit neuer Wirkstoffe zu garantieren“. Freilich seien die dabei verwendeten personenbezogenen Daten höchst sensibel und entsprechend schutzwürdig. Daher müsse die Zustimmung der jeweiligen Patienten zur Verwendung ihrer Daten eingeholt werden. Diese würden im Rahmen der klinischen Forschung pseudonymisiert bzw. anonymisiert. Nur der behandelnde Arzt könne die Verbindung zwischen den Daten und den Patienten herstellen. Ihm obliege folglich „eine hohe Verantwortung“. Wichtig ist laut Latzko die Möglichkeit, anonymisierte Daten, die im Zuge einer Studie gewonnen wurden, auch für andere Studien weiterverwenden zu können: „So können zum Beispiel Erkenntnisse aus der Krebstherapie auch für die Behandlung von Multipler Sklerose hilfreich sein.“ Das verdeutliche die Bedeutung des „Broad Consent“. Grundsätzlich hält Latzko das Datenschutzanpassungsgesetz Wissenschaft und Forschung für gut gelungen: „Es schafft eine ausgewogene Balance, indem es unter strengen Auflagen die Datennutzung für Forschungszwecke ermöglicht und gleichzeitig den notwendigen Schutz personen-bezogener Daten sicherstellt.“
Data Sharing als „unbedingtes Muss“
Michaela Fritz, Mitglied der ÖGMBT und Vizerektorin der Medizinischen Universi-tät Wien, erläuterte ihre Position bei einer Pressekonferenz des Wissenschaftsministeriums im Vorfeld der Beschlussfassung des Gesetzes. Die moderne medizinische Forschung arbeite mit anonymisierten, pseudonymisierten, aber auch personenbezogenen Daten. Und der Zugang zu diesen Daten müsse gewährleistet sein. Ferner bezeichnete sie die Weitergabe von Daten an nationale und internationale Partner sowie das Data Sharing als „unbedingtes Muss“. Fritz zufolge erstellten die österreichischen „Forscher des Jahres“ von 2017 und 2018 eine gemeinsame Publikation zu Diabetes-Erkrankungen. Dabei untersuchten sie die Daten von rund 1,8 Millionen Patienten, von denen 300.000 an Typ-2-Diabetes leiden und die mit etwa 300 verschiedenen Medikamentenkombinationen behandelt werden. Aus derartigen Big-Data-Analysen ließen sich neue und relevante Gesund-heits- und Präventionsmaßnahmen ableiten. Unter anderem stellten die Forscher in dieser Publikation fest, dass die verabreichten Medikamentenkombinationen das Krebsrisiko erhöhen oder senken.
Im Zuge der Präventionsforschung können laut Fritz auch kleinere Datenbestände (Register) relevant sein, insbesondere, wenn es darum geht, Daten von Patienten über einen längeren Zeitraum nachzuverfolgen. Leidet jemand im Kindesalter an einer Krankheit, kann es sinnvoll sein, seine Entwicklung bis ins Erwachsenenalter zu beobachten. So lässt sich möglicherweise feststellen, wie sich Umwelteinflüsse, genetische Einflüsse sowie pränatale Ereignisse auf den Krankheitsverlauf auswirken.
Hinsichtlich der Biobanken verwies Fritz darauf, dass die onkologische Forschung zunehmend versucht, die Mechanismen hinter der jeweiligen Krebserkrankung zu erkennen. Diese Mechanismen können aber auch für andere Krebsarten relevant sein. Umso wichtiger sei daher der „Broad Consent“. Und noch ein Beispiel für die Sinnhaftigkeit der neuen Regelungen führte Fritz ins Treffen: die „Seltenen Erkrankungen“. Ihr zufolge fordern davon Betroffene „ganz massiv, dass die Forschung diese Daten weitergibt und europäisch teilt. Denn nur so lassen sich neue Erkenntnisse gewinnen“. Ihrer Ansicht nach wurde mit den neuen Regelungen „ein Spagat geschafft“. Einerseits sei das Individuum geschützt. Andererseits habe die Forschung unbürokratisch Zugang zu Daten sowie Rechtssicherheit.














































